Profesor a Tiempo Completo
Departamento de Bioingeniería e Ingeniería Química
Los bacteriófagos tienen un gran potencial como sistema de expresión de componentes. Desde hace décadas se emplea a los fagos para el desarrollo de nuevos antimicrobianos, anticuerpos monoclonales y recientemente el desarrollo de nuevos biomateriales. El bacteriófago mas empleado para dicho fin es el fago filamentoso M13; cuya estructura permite combinar las proteínas de su superficie con proteínas de interés presentadas. El virus M13 tiene la capacidad de infectar a cepas de Escherichia coli cepas que muestran el F-pilus [1]. Posee ADN de cadena sencilla con una cápside compuesta por la proteína PVIII y cuatro proteínas de cubierta menores (PIII, PVI, PVII y PIX). Posee 2700 copias de la proteína PVIII que conforman el cilindro principal, y cinco copias de la proteínas PIII y PVI y PVII y PIX en sus extremos. Las proteínas PVIII y PIII son las más comúnmente empleadas para realizar el procedimiento conocido como despliegue de bacteriófagos o “phage display” [1,2].
El desarrollo de anticuerpos depende de una cuidadosa selección de la afinidad antígeno/anticuerpo. Usualmente una biblioteca de variantes de anticuerpos se inserta genéticamente al vector conteniendo el genoma viral en donde se fusiona la proteína gp3 (P III) de la cubierta del bacteriófago M13 al péptido o anticuerpo. La selección in vitro permite realizar rondas de filtración de bacteriófagos para seleccionar aquellos que poseen una alta afinidad por la molécula blanco (antígeno). La selección se produce mediante el “biopanning”; una unión in vitro antígeno-anticuerpo empleando el método de ELISA, “phage display”[1-3].
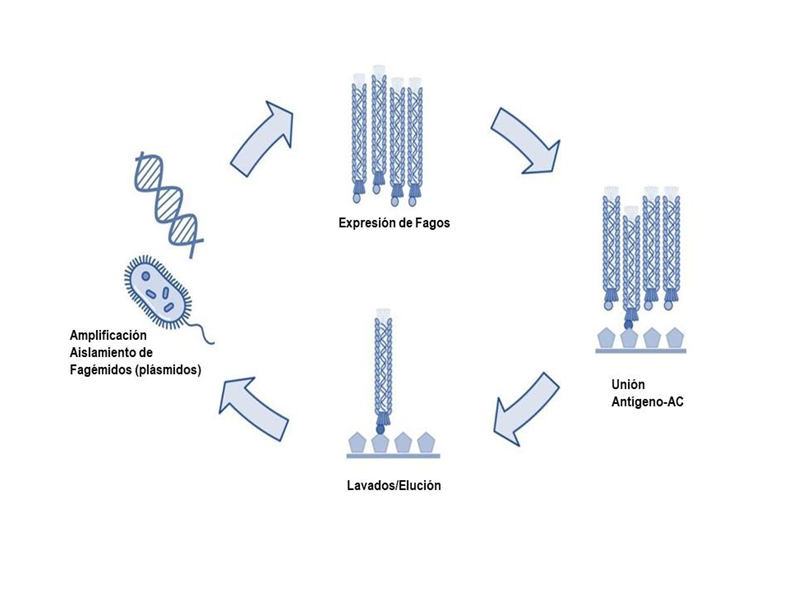
La eficiente selección depende de la alta afinidad del péptido (antígeno) adherido a la placa de ELISA y el anticuerpo fusionado a PIII y parte de la estructura de la cubierta del fago M13. En la Figura 1 se muestra el procedimiento “biopanning” de cuatro etapas. Inicialmente, la creación del fagémido por tanto la biblioteca que permite expresar las variantes de anticuerpos en el virus M13. La segunda etapa el procedimiento de selección mediante un ELISA, en donde una péptido o antígeno se inmoviliza en la placa a la cual se exponen los bacteriófagos purificados. El siguiente proceso de lavados permite descartar a aquellos que poseen una baja o nula afinidad mediante una elución. Los bacteriófagos eludidos son amplificados en Escherichia coli, para recuperar el material genético mediante una extracción de plásmidos. Los plásmidos (famégidos) aislados se emplean para realizar una siguiente ronda en el proceso o “biopanning”. Este procedimiento se realizar 2 – 4 veces para lograr encontrar anticuerpos con una alta afinidad [2,3].
Los procedimientos anteriores son laboriosos y no se produce un monitoreo cuantitativo de la afinidad antígeno-anticuerpo en el proceso. Idealmente, el proceso de selección de nuevos anticuerpos se beneficiaria de un monitoreo en cada ronda de “panning”; de modo que permita reducir costos y concentrar esfuerzos en las moléculas con alta afinidad y más prometedoras [1].
Se propone emplear la tecnología de “pegamento molecular” para acoplarla al sistema de despliegue de bacteriófagos. Los péptidos que realizan bionconjugación en la naturaleza tienen la capacidad de unir covalentemente dos proteínas. Un ejemplo son los péptidos SpyTag y SpyCatcher los cuales reaccionan espontáneamente mediante sus residuos de aspártico y lisina, respectivamente [2, 3]. Aprovechando al pegamento molecular se puede fusionar proteínas como anticuerpos con la estructura del bacteriófago M13. La propuesta conocida como “spy-display” que se describe en la Figura 2 [3], permite fusionar posterior a la expresión de la biblioteca a la proteína PIII con el anticuerpo mediada por SpyTag/SpyCatcher. Lo anterior ofrece las siguientes ventajas: A) Se evitan los inconvenientes de ensamblaje de fago M13 por tamaños excesivo de proteínas fusionadas a PIII. B) El plásmido fagemido solo contiene las proteínas del M13, mejorando la producción viral. Mientas que un segundo plásmido de expresión se emplea para la expresión de los anticuerpos. C) Se pueden analizar anticuerpos de tamaño mayor a los de cadena sencilla. D) El sistema es modular y se podría añadir un fluoróforo, proteína biomarcador o enzima para monitorear la señal de la unión de los anticuerpos para decidir específicamente que población emplear para realizar la amplificación [3].
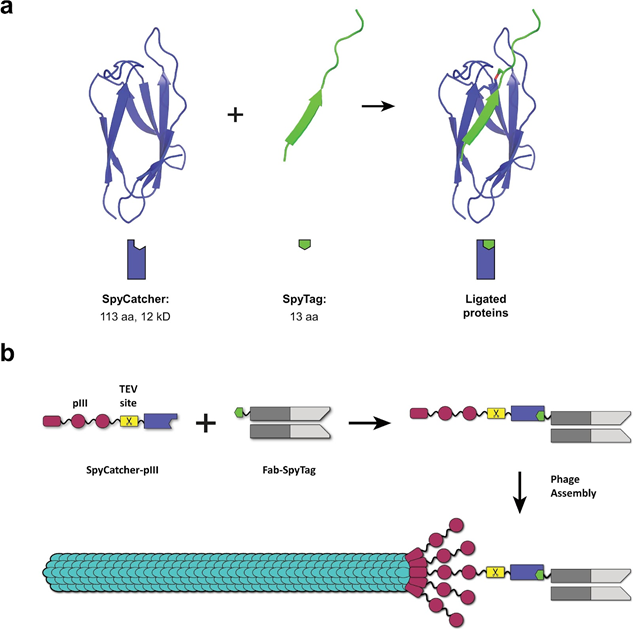
Frecuentemente, durante la selección in vitro de biomoléculas se obtiene falsos positivos. Lo anterior eleva los costos y reduce la eficiencia de los métodos. En el caso del despliegue de anticuerpos, las bibliotecas poseen muchas veces versiones truncadas que se produce durante la maduración del fago M13 en la membrana. El sistema planteado Spy-display permite una producción y maduración intacta del virus M13 produciendo una versión “wild type” la cual es fusionada post-traduccionalmente a la biblioteca de anticuerpos. Se obtiene un población viral mas robusta y niveles de infección mayores. Por lo tanto, el sistema muestra una prometedora manera de identificar nuevos anticuerpos y diseñar variantes de anticuerpos o sintéticos que pueden representar una contribución para pruebas de terapias mas eficaces o ensayos de detección rápida de virus en ensayos de bajo costo [2-4].
Referencias
1.- Passaretti, P., Khan, I., Dafforn, T.R. et al. Improvements in the production of purified M13 bacteriophage bio-nanoparticle. Sci Rep 10, 18538 (2020). https://doi.org/10.1038/s41598-020-75205-3
2.- Fierle, J.K., Abram-Saliba, J., Brioschi, M. et al. Integrating SpyCatcher/SpyTag covalent fusion technology into phage display workflows for rapid antibody discovery. Sci Rep 9, 12815 (2019). https://doi.org/10.1038/s41598-019-49233-7
3.- Kellmann SJ, Hentrich C, Putyrski M, Hanuschka H, Cavada M, Knappik A, Ylera F. SpyDisplay: A versatile phage display selection system using SpyTag/SpyCatcher technology. MAbs. 2023 Jan-Dec;15(1):2177978. doi: 10.1080/19420862.2023.2177978
4.- Qi H, Lu H, Qiu HJ, Petrenko V, Liu A. Phagemid vectors for phage display: properties, characteristics and construction. J Mol Biol. 2012;417(3):129–43. doi: 10.1016/j.jmb.2012.01.038.