Profesor a Tiempo Completo
Departamento de Bioingeniería e Ingeniería Química
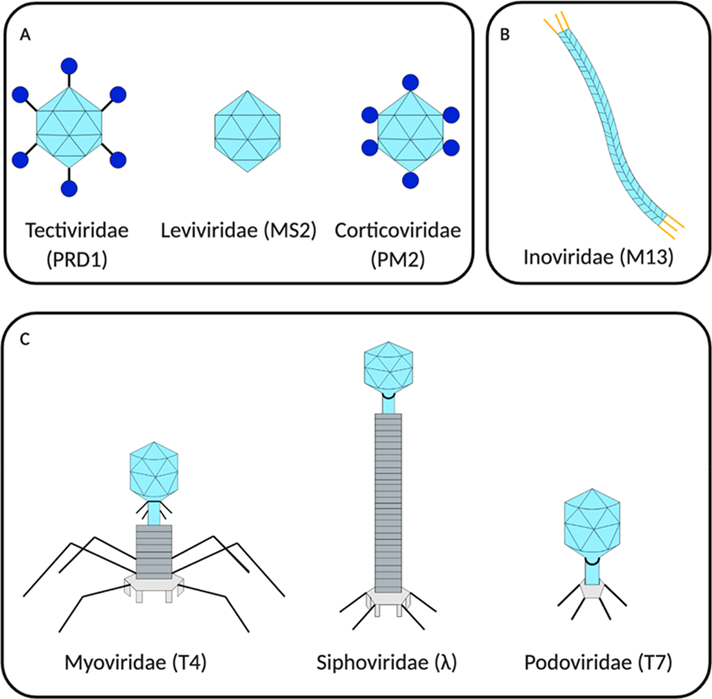
Los bacteriófagos son virus que infectan bacterias que se pueden encontrar en todos los ecosistemas del planeta. La infección les permite utilizar la maquinaria de transcripción bacteriana para replicarse. Al interior de su cápside proteica, los bacteriófagos almacenan su genoma compuesto por ADN o ARN con la información para codificar proteínas estructurales virales. La cápside puede ser icosaédrico (cortico virus), filamentoso (fago M13) o en forma cabeza-cola (fago T7) (Figura 1). Los bacteriófagos se unen a la membrana extracelular bacteriana para introducir su genoma en el citoplasma bacteriano. Se conocen dos ciclos de vida conocidos como infección lítica o lisogénica. En el ciclo de vida lítico, los bacteriófagos inducen una muerte rápida de las bacterias y al mismo tiempo se liberan cientos de nuevos virus. En cambio, en el ciclo de vida lisogénico, los bacteriófagos se reproducen al mismo tiempo que las bacterias, sin destruirlas [1].
Los bacteriófagos han llegado a ser una herramienta importante como un modelo de estudio, para el entendimiento de la biología. Su estudio permitió conocer que el ADN es el material genético, que un codón codifica para un aminoácido, y dilucidar los procesos de regulación génica. En el campo de la biología molecular, la clonación de genes a través del ensamblaje de ADN ha sido posible al combinar enzimas de restricción que las bacterias usan para cortar secuencias específicas de ADN junto con el proceso de unir moléculas de ADN con la ADN ligasa del bacteriófago T4 [1, 2]. Asimismo, las ADN polimerasas de fagos han servido para la secuenciación de moléculas de ADN [2]. Otra tecnología en pleno auge para la edición genética es CRISPR-Cas (“clustered regularly interspaced short palindromic repeats”), lal cual está basada en el mecanismo de defensa de bacterias contra bacteriófagos. En las aplicaciones para la Bioingeniería, la modificación genética del genoma de estos virus ha permitido modificar las proteínas de su superficie generando nuevos nanomateriales para el diagnóstico de cáncer, agentes terapéuticos contra bacterias multidrogorresistentes. La plataforma tecnológica de librerías de expresión en fagos (phage display) es un sistema muy utilizado en la industria farmacéutica para evaluar anticuerpos, antígenos y toxinas relacionadas a cáncer. En este sistema algunas proteínas de la cápside viral están fusionadas a péptidos con afinidad a células eucariotas, lo que ha permitido la liberación de medicamentos a células de cancerosas y el desarrollo de vacunas basadas en fagos al fusionar bacteriófagos con antígenos de patógenos humanos. Existen esfuerzos para implementar una vacuna con el ántrax utilizando como sistema de entrega del antígeno PA, al bacteriófago T4 [3, 4].
En la industria alimentaria, los bacteriófagos se han usado como agentes biosanitizantes al matar bacterias patógenas (Campylobacter jejuni y Listeria monocytogenes) o produciendo enzimas (depolimerasas y endolisinas) para reducir la formación de biopelículas en las superficies de los materiales de uso industrial. Otro enfoque ha sido la aplicación de bacteriofagos como agente biopreservante para alargar el tiempo de vida en productos alimenticios al matar bacterias responsables en la putrefacción de los alimentos. En la industria láctea, los bacteriofagos pueden eliminar patógenos como Staphylococcus.
En la agricultura, los bacteriofagos se han usado como agentes para combatir infecciones a plantas como Erwinia amylovora que infecta a la manzana, Ralstonia solanacearum y Xanthomonas campestri que infectan al tomate [5].
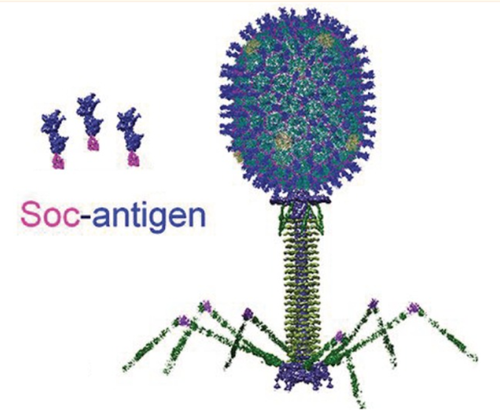
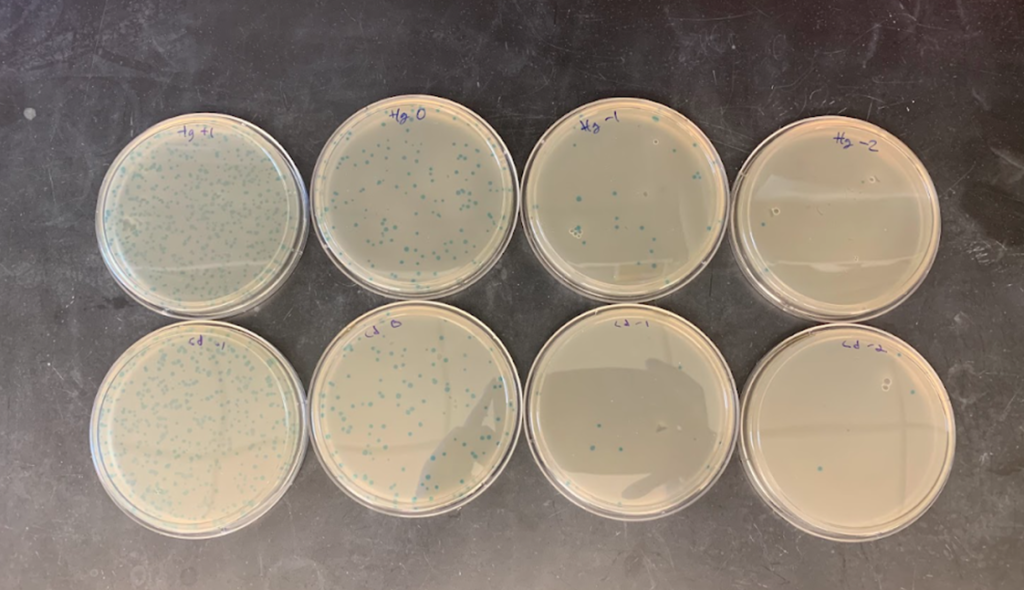
En el Departamento de Bioingeniería y Centro BIO de la UTEC, nos encontramos desarrollando un proyecto en colaboración con el MIT (USA); para la remoción de metales pesados en el medio ambiente empleando bacteriófagos genéticamente modificados. Idealmente, se desea implementar hidrogeles capaces de contener estos virus con una alta capacidad de absorción por metales pesados para ser combinados con agua contaminada. En la imagen, se observan los primeros ensayos de construcción de bacteriófagos obtenidos con afinidad a mercurio y cadmio, desarrollado por UTEC y el MIT (Figura 3).
Referencias
[1] A. B. Monk, C. D. Rees, P. Barrow, S. Hagens, and D. R. Harper, “Bacteriophage applications: where are we now?,” Lett. Appl. Microbiol., vol. 51, no. 4, pp. 363–369, Oct. 2010, doi: 10.1111/j.1472-765X.2010.02916.x.
[2] M. Mahler, A. R. Costa, S. P. B. van Beljouw, P. C. Fineran, and S. J. J. Brouns, “Approaches for bacteriophage genome engineering,” Trends Biotechnol., vol. 41, no. 5, pp. 669–685, May 2023, doi: 10.1016/j.tibtech.2022.08.008.
[3] Tao P, Mahalingam M, Zhu J, et al. A bacteriophage T4 nanoparticle‐based dual vaccine against anthrax and plague. mBio. 2018;9(5):e01926‐e01918. 10.1128/mBio.01926-18.
[4] Hess KL, Jewell CM. Phage display as a tool for vaccine and immunotherapy development. Bioeng Transl Med. 2019 Sep 18;5(1):e10142. doi: 10.1002/btm2.10142. PMID: 31989033; PMCID: PMC6971447.
[5] M. Połaska and B. Sokołowska, “Bacteriophages—a new hope or a huge problem in the food industry,” AIMS Microbiology, vol. 5, no. 4, p. 324, 2019, doi: 10.3934/microbiol.2019.4.324.